library(tidyverse)
# fixe la graine à 105
set.seed(105)La loi normale
Introduction
Les lois de proababilités
Une loi de probabilité revient à prévoir le comportement d’une expérience aléatoire. Les grandes lois de probabilité sont basées sur le fonctionnement de jeu de hasard et ont été ensuite formalisées.
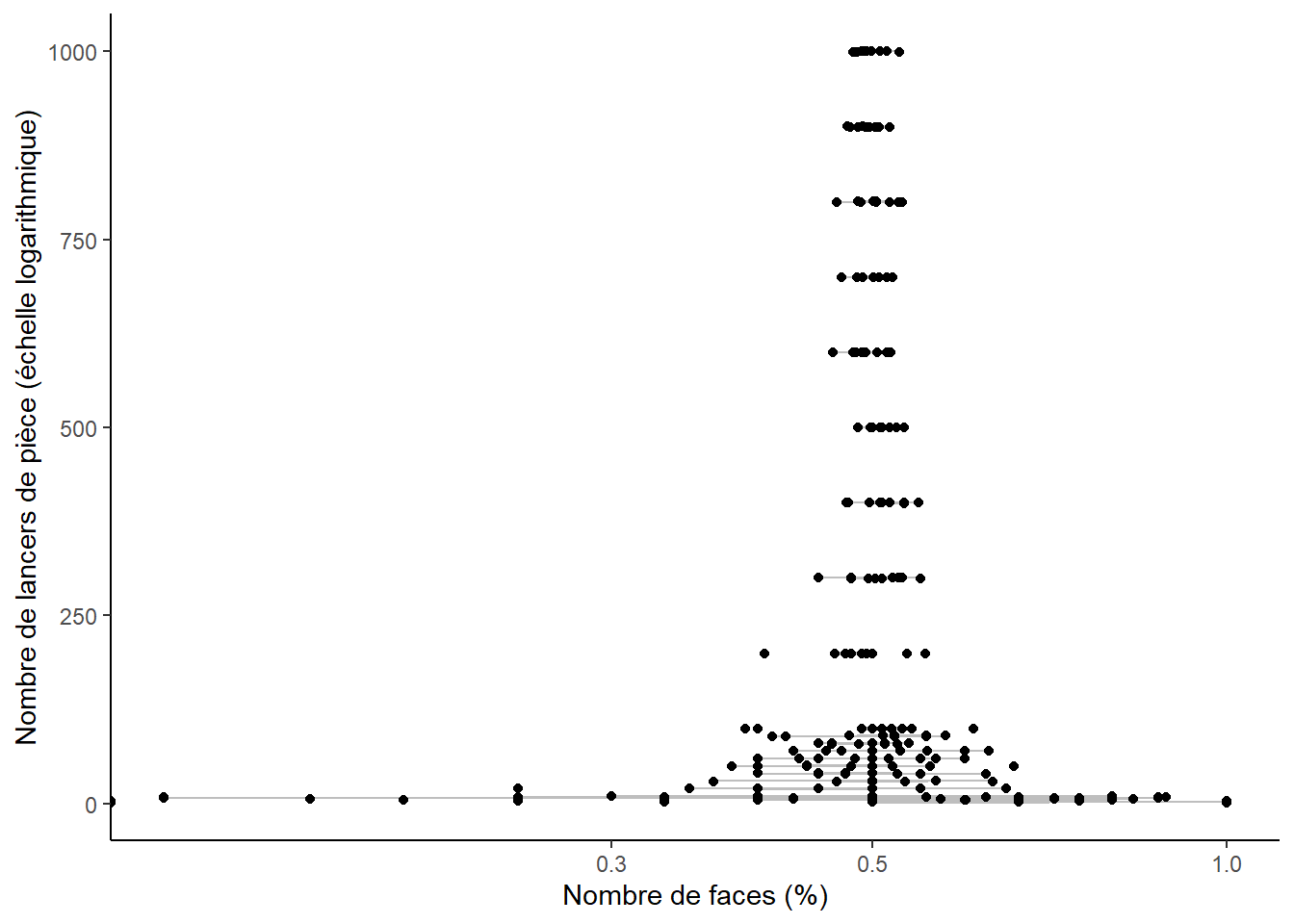
Naturellement, tout le monde comprend que lorsqu’on joue à lancer une pièce (équilibrée) au hasard, chacune des faces à autant de chance de sortir.
Il est impossible de prévoir le nombre de piles et de faces pour un petit nombre de lancers, par exemple six.
Par contre, plus ce nombre augmente, plus on peut prévoir le nombre de fois que le côté face va sortir, car le nombre de lancers équilibre le résultat.
# création d'un vecteur contenant le nombre de lancers
nb_lancers <- c(
1:9, # 1, 2, 3... 9
seq(from = 10, to = 100, by = 10), # 10, 20... 100
seq(from = 200, to = 1000, by = 100) # 200, 300... 1000
)
# 10 simulations pour chaque valeurs de nb_lancers de pièces
purrr::map(
c(1:10),
~ bind_rows(
bind_cols(
nb_lancers = nb_lancers,
nb_lancers %>%
purrr::map(
~ rbinom(.x, 1, 0.5)
) %>%
purrr::map(sum) %>%
purrr::map(as_tibble) %>%
bind_rows() %>%
rename(nb_faces = value)
)
)
) %>%
bind_rows() %>% # mise en commun des lignes
mutate(
pourcentage_face = nb_faces /nb_lancers # calcul du pourcentage de réussites
) %>% # création du graphique
ggplot() +
aes(y = nb_lancers, x = pourcentage_face, group = nb_lancers) +
geom_boxplot(color = "grey") + # ajout des boîtes à moustaches
geom_jitter() + # ajout des points
scale_x_log10() + # choix d'une échelle logarthmique
theme_classic() +
labs(
y = "Nombre de lancers de pièce (échelle logarithmique)",
x = "Nombre de faces (%)"
)
Origine de la loi normale
Si la loi de probabilité uniforme est la plus intuitive, la loi de probabilité normal est la plus couramment utilisée. En effet, c’est celle qui est la plus proche des phénomènes biologiques.
Contrairement aux lois binomiales et de Poisson, la loi normale est une loi de probabilité continue définie par deux paramètres : son espérance (µ) et sa variance (σ). Sa densité de probabilité s’écrit comme suit.
Si X ∽ Ν(μ,σ²) alors \[ f(x)=\frac{1}{(σ√2π)}{e^{\frac{-1}{2} ({\frac{x-μ}{σ})^2}}} \] pour tous \[ x ∈ {R^+} \]
C’est une densité de probabilité et non une fonction de masse qui permet de la définir.
La densité de probabilité est symétrique, le centre étant l’espérance, l’écart-type la largeur du pic comme visible sur la figure.
# choix de l'intervalle de représentation
intervalle <- seq(-15, 15, 0.1)
# loi normale de paramètres µ égale à -5, -3, 0 ou 3 et σ égale à 2, 0.8, 1 ou 3
tibble(
loi_normale =
rep(
c(
"µ = 0 & \U03B4 = 1",
"µ = 3 & \U03B4 = 3",
"µ = -3 & \U03B4 = 0.8",
"µ = -5 & \U03B4 = 2"
),
each = length(intervalle)
),
x = rep(intervalle, 4),
densite =
c(
dnorm(intervalle, mean = 0, sd = 1),
dnorm(intervalle, mean = 3, sd = 3),
dnorm(intervalle, mean = -3, sd = 0.8),
dnorm(intervalle, mean = -5, sd = 2))
) %>%
ggplot() +
aes(x = x, y = densite, color = loi_normale) +
geom_line(size = 1) +
theme_classic() +
scale_color_manual(values = c("blue", "dark red", "black", "gold")) +
ylab("densite de probabilité (f(x))") +
labs(color = "Loi normale")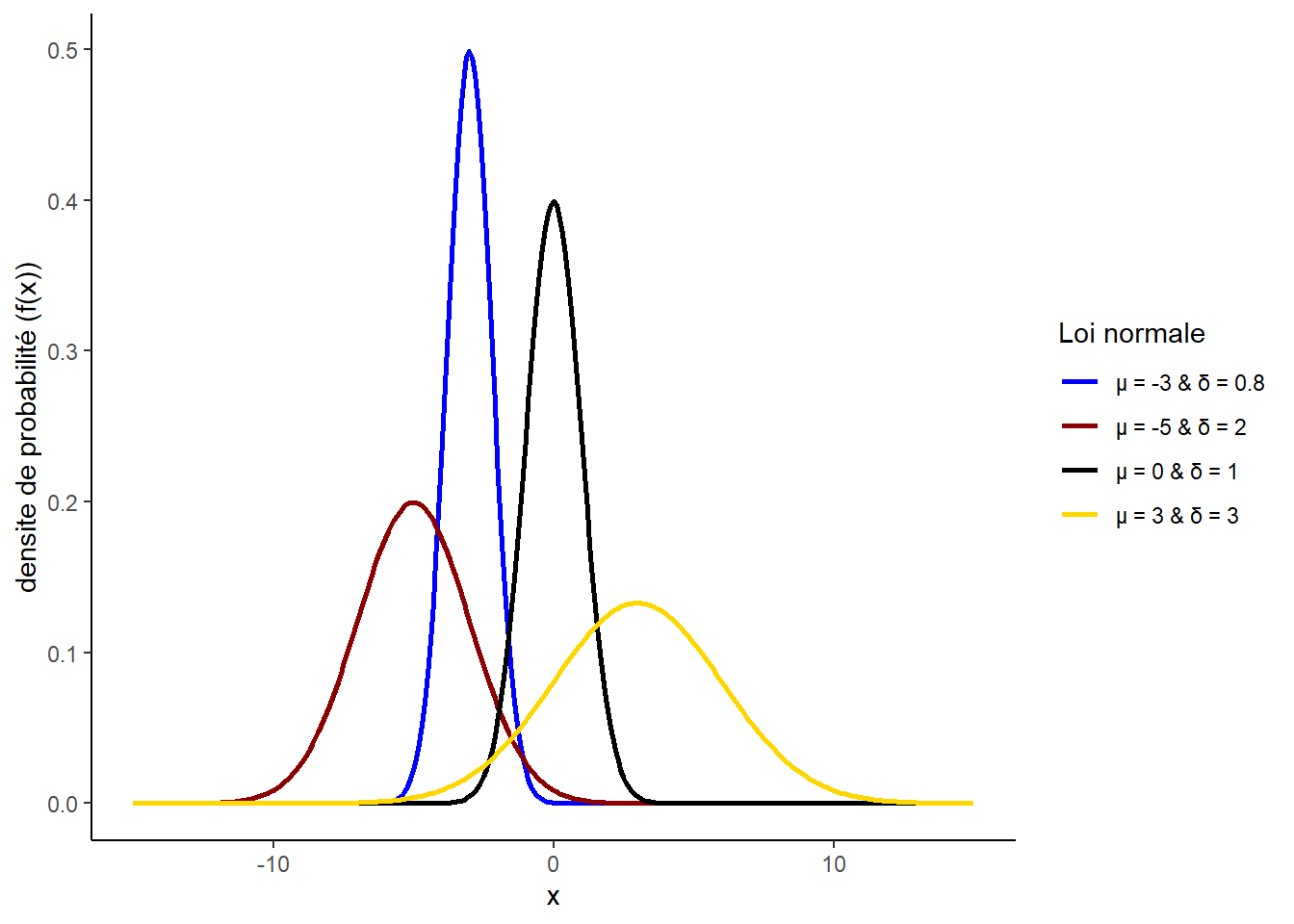
ggsave("img/loi_normale.png")Approximation
Les évènements aléatoires quel qu’ils soient, rendent impossible la prédiction exacte.
Cela signifie que quand on rejette à 5%, dans 5 cas sur 100, c’est vrai mais on le rejette quand même !
# choix de l'intervalle de représentation
intervalle <- seq(-5, 5, 0.1)
# test bilatéral
bilateral <- tibble(
x = intervalle,
"densité" = dnorm(intervalle, mean = 0, sd = 1),
region =
case_when(
x < qnorm(0.025) ~ "région critique basse",
x < qnorm(0.975) ~ "région de confiance",
TRUE ~ "région critique haute"
)
) %>%
ggplot() +
aes(x = x, y = `densité`) +
geom_line(size = 1) +
geom_area(aes(fill = region)) +
scale_fill_manual(values = c("blue", "red", "green")) +
geom_vline(xintercept = qnorm(0.025), size = 1, linetype = "dashed") +
geom_vline(xintercept = qnorm(0.975), size = 1, linetype = "dashed") +
geom_text(aes(x = -4, y = 0.06, label = "2,5 % des valeurs")) +
geom_text(aes(x = 4, y = 0.06, label = "2,5 % des valeurs")) +
geom_text(aes(x = 0, y = 0.42, label = "95 % des valeurs")) +
theme_classic()
# test unilatéral à gauche
unilateral_gauche <- tibble(
x = intervalle,
"densité" = dnorm(intervalle, mean = 0, sd = 1),
region =
case_when(
x < qnorm(0.05) ~ "région critique basse",
TRUE ~ "région de confiance"
)
) %>%
ggplot() +
aes(x = x, y = `densité`) +
geom_line(size = 1) +
geom_area(aes(fill = region)) +
scale_fill_manual(values = c("blue", "green")) +
geom_vline(xintercept = qnorm(0.05), size = 1, linetype = "dashed") +
geom_text(aes(x = -3.5, y = 0.06, label = "5 % des valeurs"), size = 2) +
geom_text(aes(x = 0, y = 0.42, label = "95 % des valeurs"), size = 2) +
theme_classic() +
theme(legend.position = "none")
# test unilatéral à droite
unilateral_droit <- tibble(
x = intervalle,
"densité" = dnorm(intervalle, mean = 0, sd = 1),
region =
case_when(
x > qnorm(0.95) ~ "région critique haute",
TRUE ~ "région de confiance"
)
) %>%
ggplot() +
aes(x = x, y = `densité`) +
geom_line(size = 1) +
geom_area(aes(fill = region)) +
scale_fill_manual(values = c("red", "green")) +
geom_vline(xintercept = qnorm(0.95), size = 1, linetype = "dashed") +
geom_text(aes(x = 3.5, y = 0.06, label = "5 % des valeurs"), size = 2) +
geom_text(aes(x = 0, y = 0.42, label = "95 % des valeurs"), size = 2) +
theme_classic() +
theme(legend.position = "none")
library(cowplot)
ggdraw() +
draw_plot(bilateral, 0, .5, 1, .5) +
draw_plot(unilateral_gauche, 0, 0, .5, .5) +
draw_plot(unilateral_droit, .5, 0, .5, .5)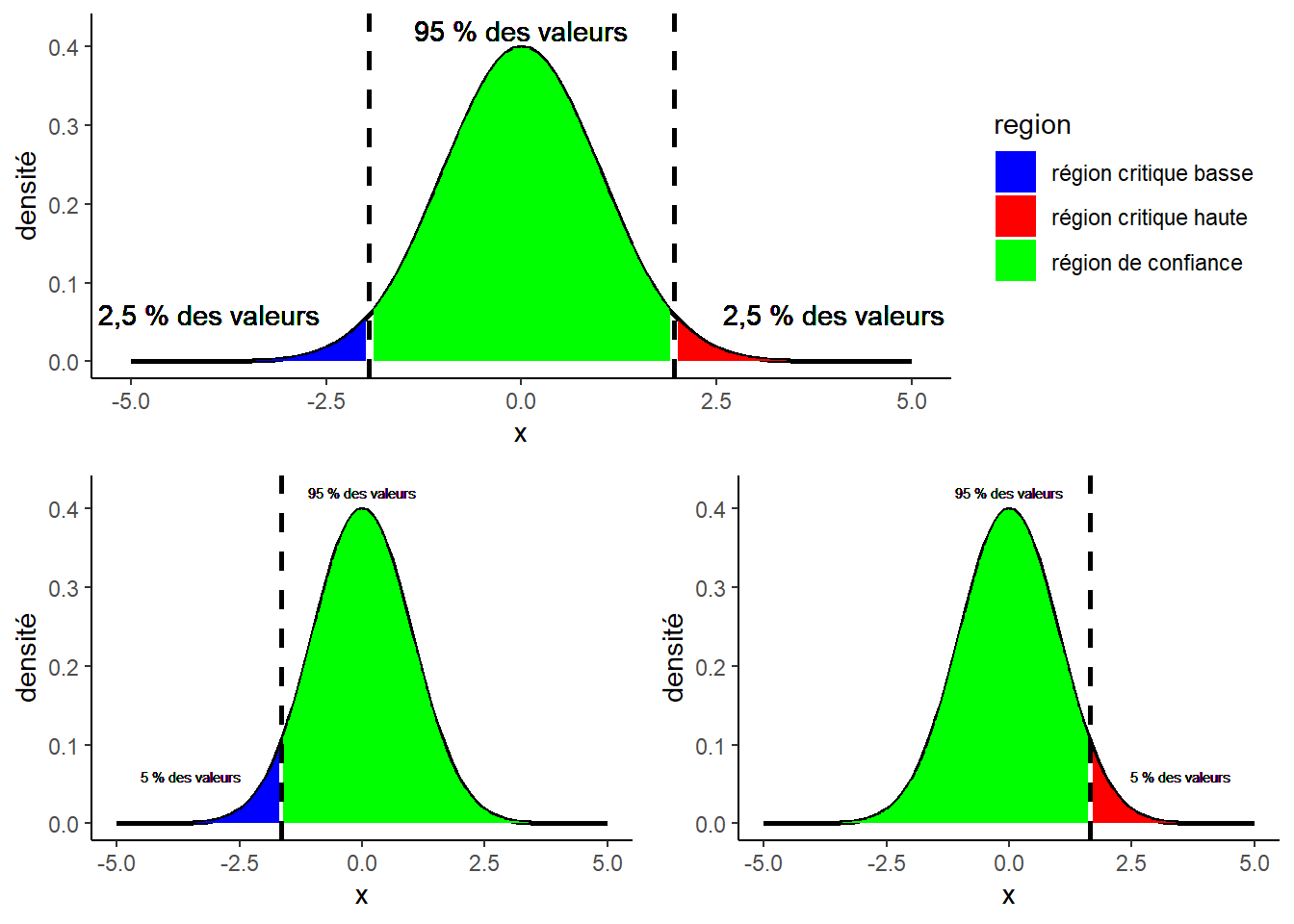
Sur les graphiques les parties rouges et bleues sont vraies mais on les rejette.
Utilité de la loi normale
Un grand nombre de tests est basé sur des données suivant une loi normale : ACP, modèle linéaire, ANOVA…
Si les données ne suivent pas de loi normale il ne faut pas faire ses tests !
Iris : jeu de données utilisé
Iris est un jeu de données avec les longueurs et largeurs de pétales et sépales mesurées sur 150 fleurs d’iris selon trois espèces (50 fleurs par espèce).
iris %>% View()
iris %>%
ggplot() +
aes(x = Petal.Length) +
geom_histogram()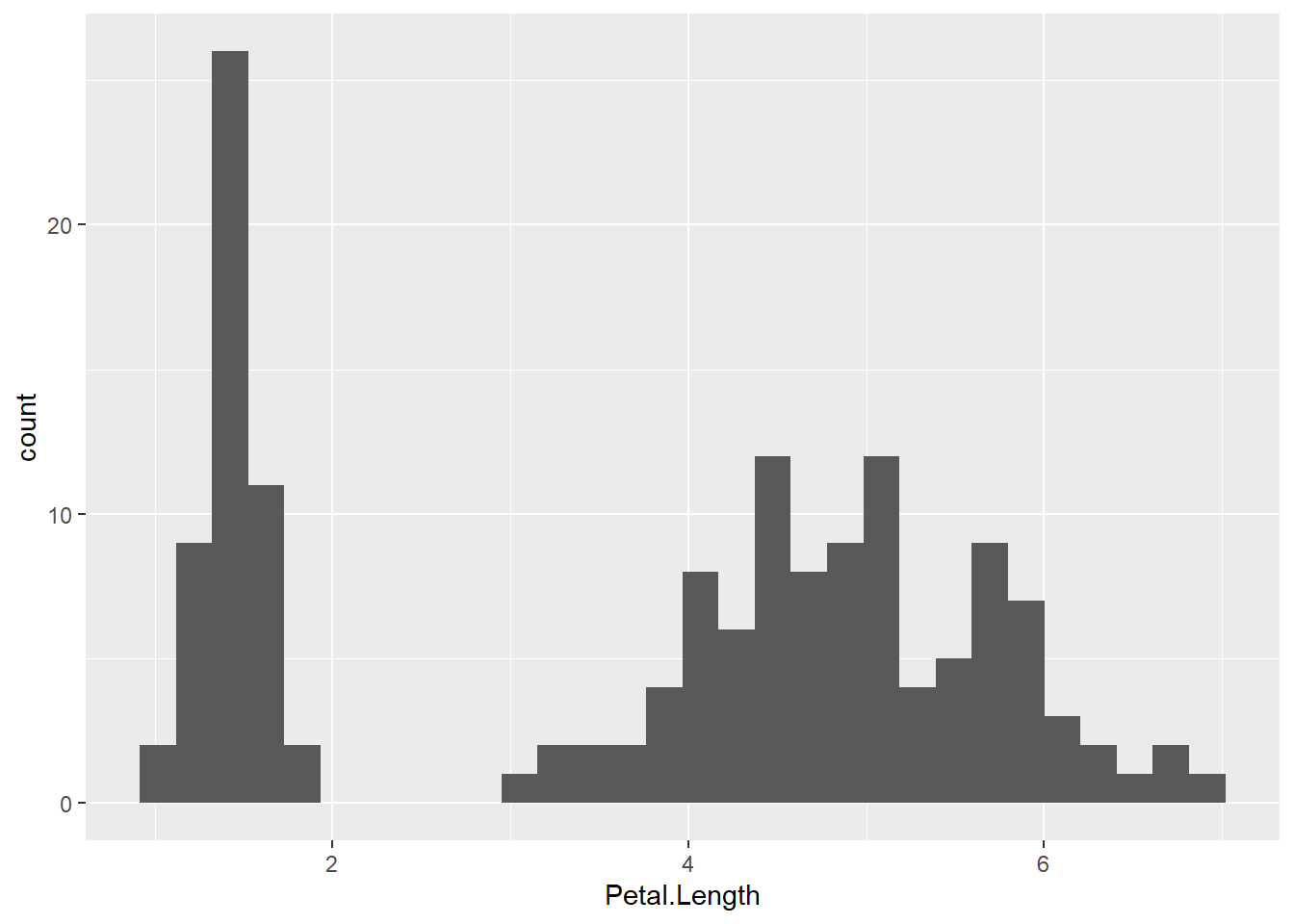
Le test de Shapiro-Wilk
Le test d’hypohtèses
1ère étape : Définir les hypothèses
Hypothèse nulle, H0 : les données suivent une loi normale
Hypothèse alternative, H1 : les données ne suivent pas une loi normale
2ème étape : Formaliser mathématiquement les hypothèses
H0 : X -> N(µ, σ²)
H1 : X –>- N(µ, σ²)
3ème étape : Tester si l’hypothèse nulle est probable ou doit-être rejetée
Si la p-valeur > 0.05, H0 n’est pas rejeté donc les données suivent une loi normale (à vérifier graphiquement)
Sinon les données ne suivent pas une loi normale.
La statistique de test
\[ {\displaystyle W= {\left (\sum \limits _ {i=1}^ {n}a_ {i}x_ { (i)}\right)^ {2} \over \sum \limits _ {i=1}^ {n} (x_ {i}- {\overline {x}})^ {2}}} \]
Réalisation du test
Le test de Shapiro-Wilk ne peut pas être utilisé quand il y a plus de 5000 valeurs.
Il doit être utilisé sur un vecteur et non sur une table entière.
shapiro.test(rnorm(n = 6000, mean = 5, sd = 3))Error in shapiro.test(rnorm(n = 6000, mean = 5, sd = 3)): la taille de l'échantillon doit être comprise entre 3 et 5000shapiro.test(
iris$Petal.Length
)
Shapiro-Wilk normality test
data: iris$Petal.Length
W = 0.87627, p-value = 7.412e-10ggplot(iris) +
aes(x = Petal.Length, fill = Species) +
geom_histogram(bins = 35)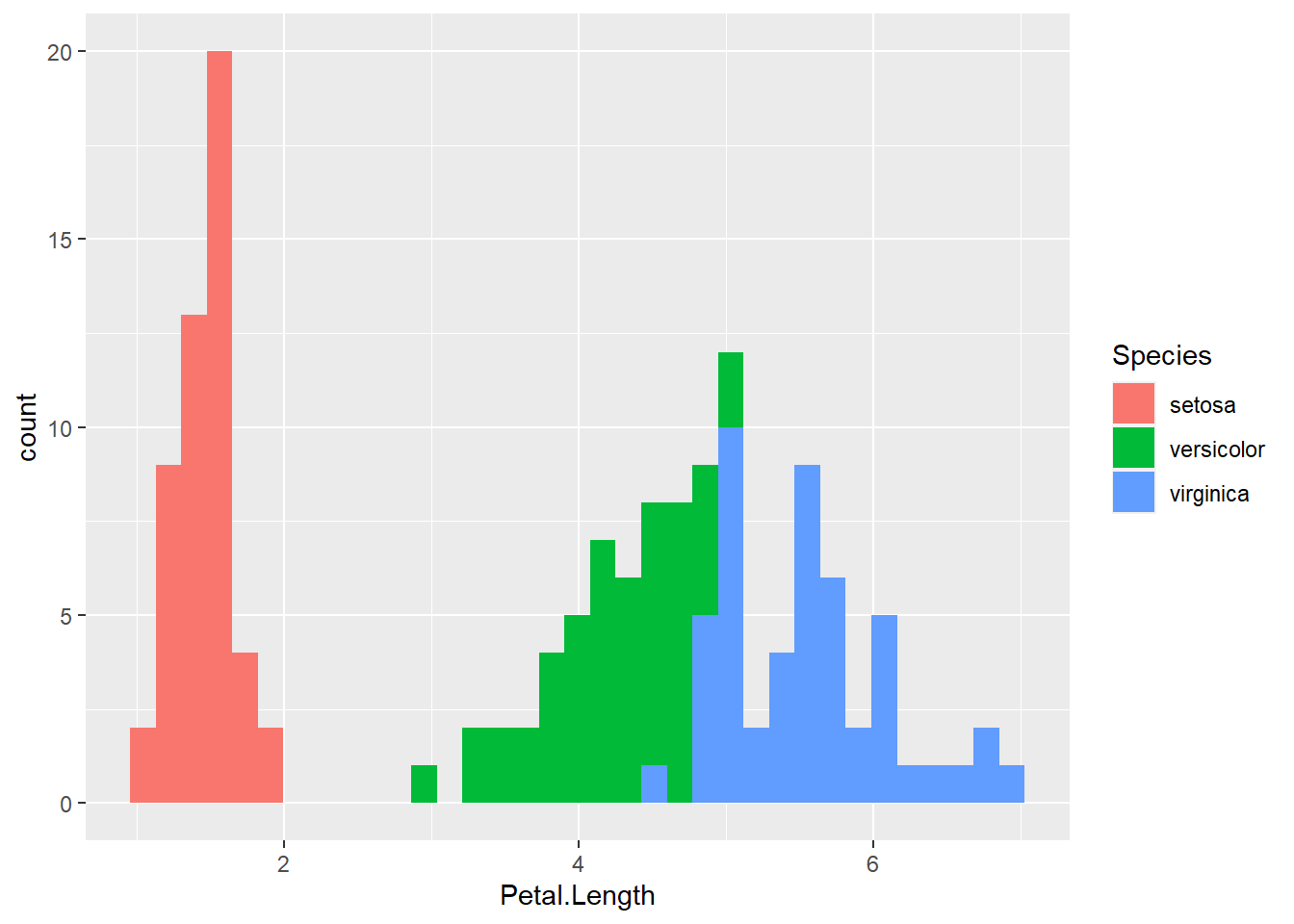
shapiro.test((iris %>% filter(Species == "setosa"))$Petal.Length)
Shapiro-Wilk normality test
data: (iris %>% filter(Species == "setosa"))$Petal.Length
W = 0.95498, p-value = 0.05481shapiro.test((iris %>% filter(Species == "versicolor"))$Petal.Length)
Shapiro-Wilk normality test
data: (iris %>% filter(Species == "versicolor"))$Petal.Length
W = 0.966, p-value = 0.1585iris %>%
filter(Species == "setosa") %>%
ggplot(aes(x = Petal.Length)) +
geom_histogram()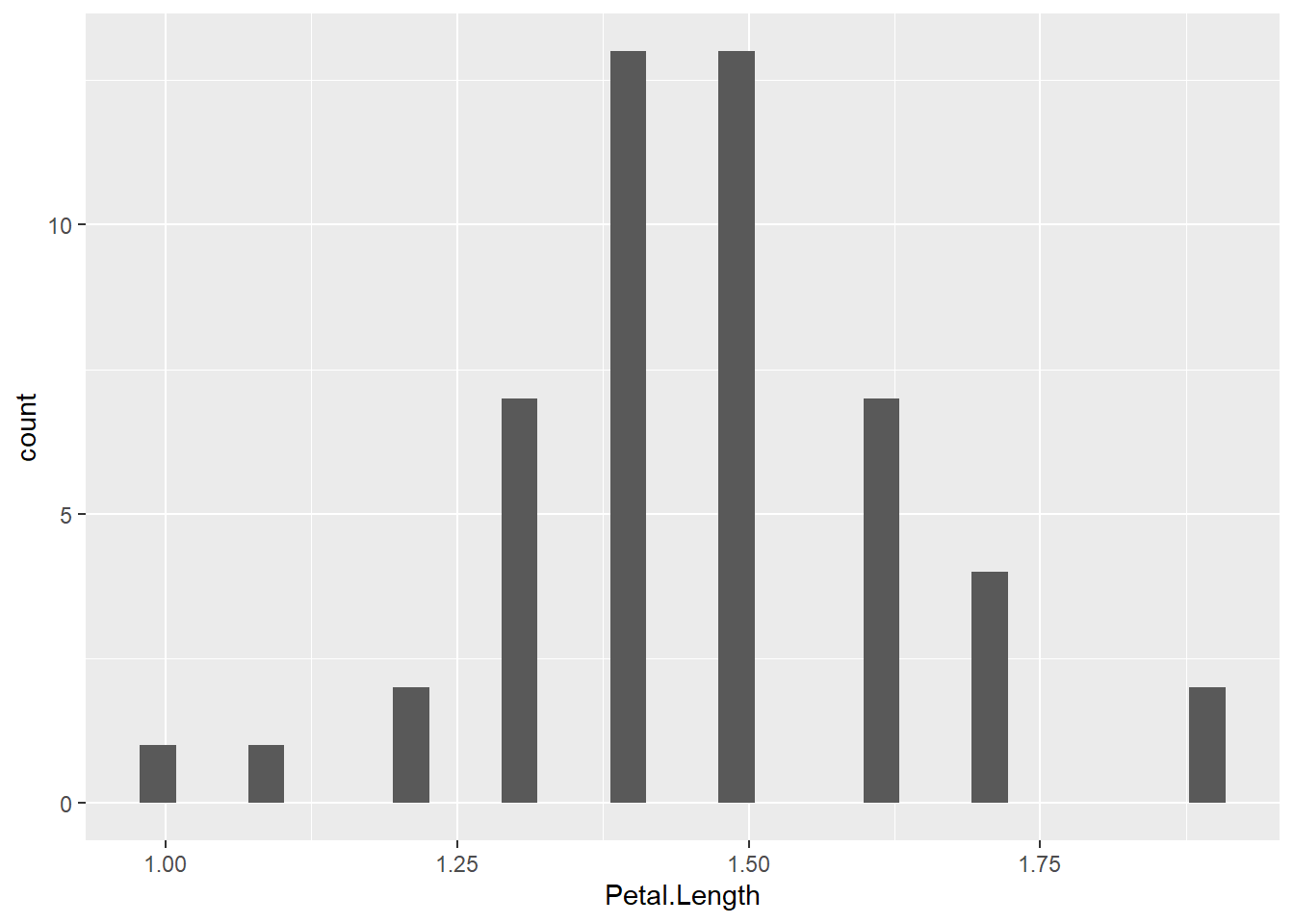
iris %>%
filter(Species == "versicolor") %>%
ggplot(aes(x = Petal.Length)) +
geom_histogram()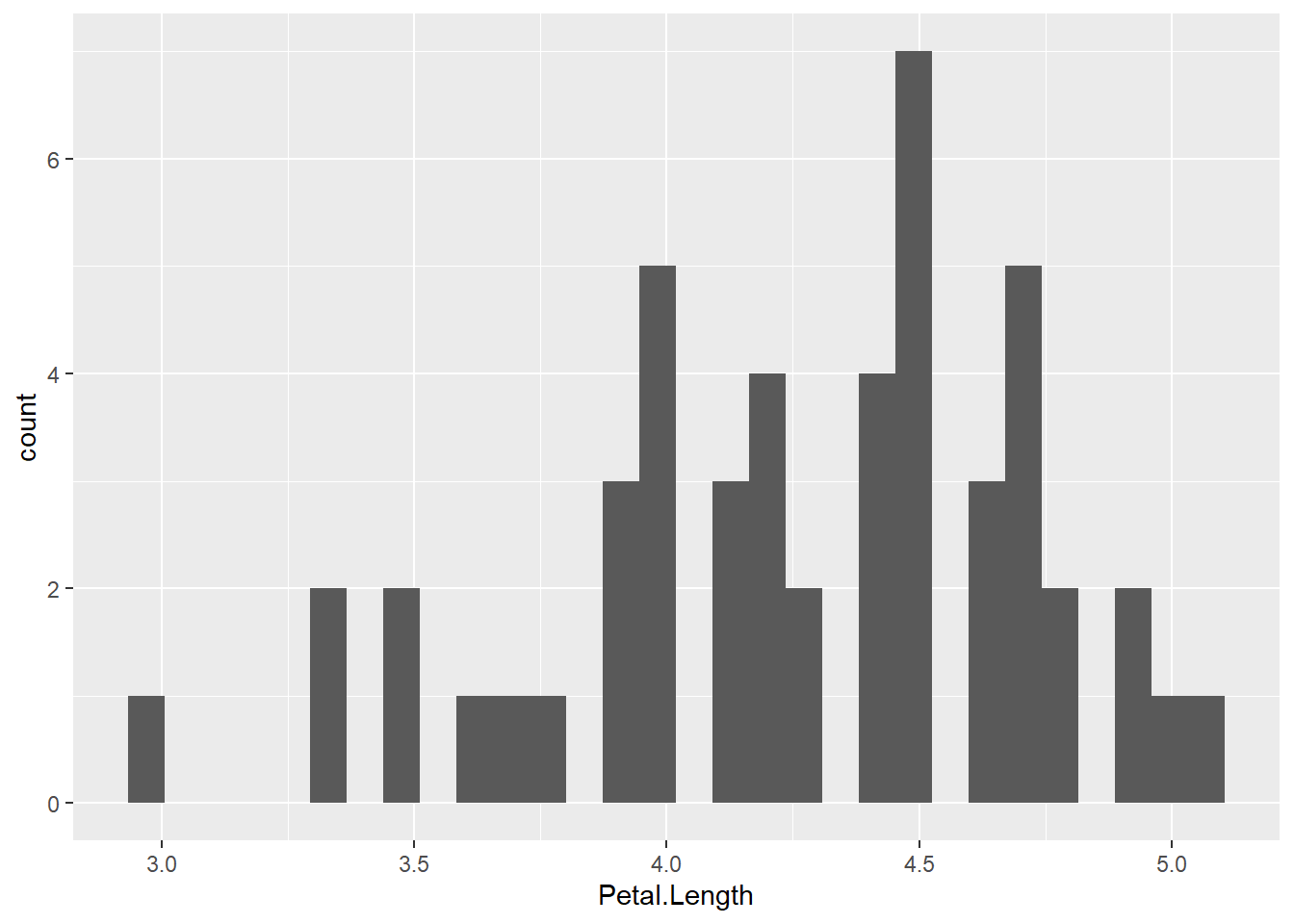
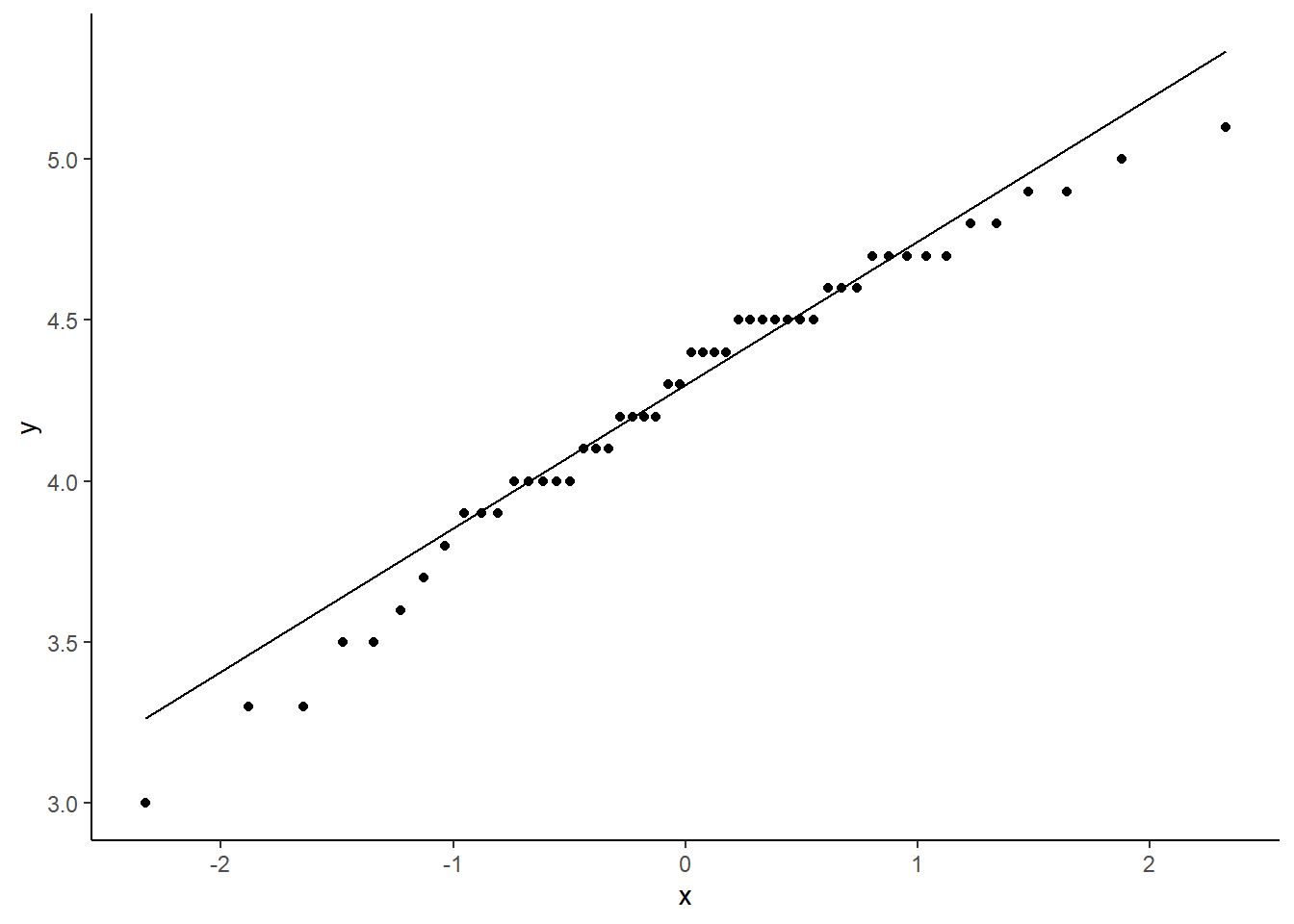
QQplot
Objectif du QQplot
Tester la normalité des données graphiquement en représentant les données réelles et celles attendus selon une loi normale.
Réalisation du graphique
iris %>%
ggplot() +
aes(sample = Petal.Length) +
geom_qq() +
geom_qq_line(color = "dark red", size = 1) +
theme_classic()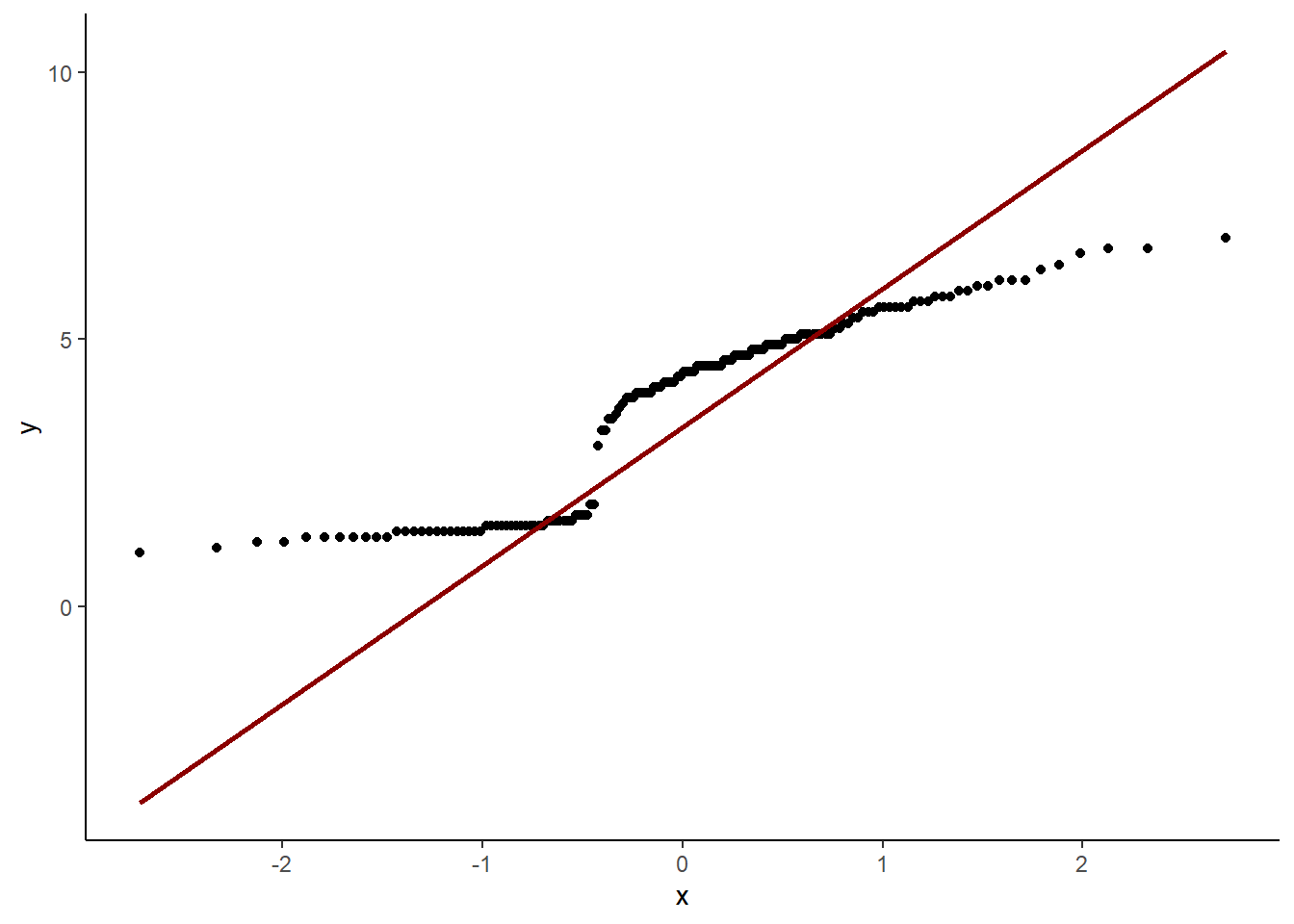
iris %>%
filter(Species == "setosa") %>%
ggplot() +
aes(sample = Petal.Length) +
geom_qq() +
geom_qq_line(color = "dark red", size = 1) +
theme_classic()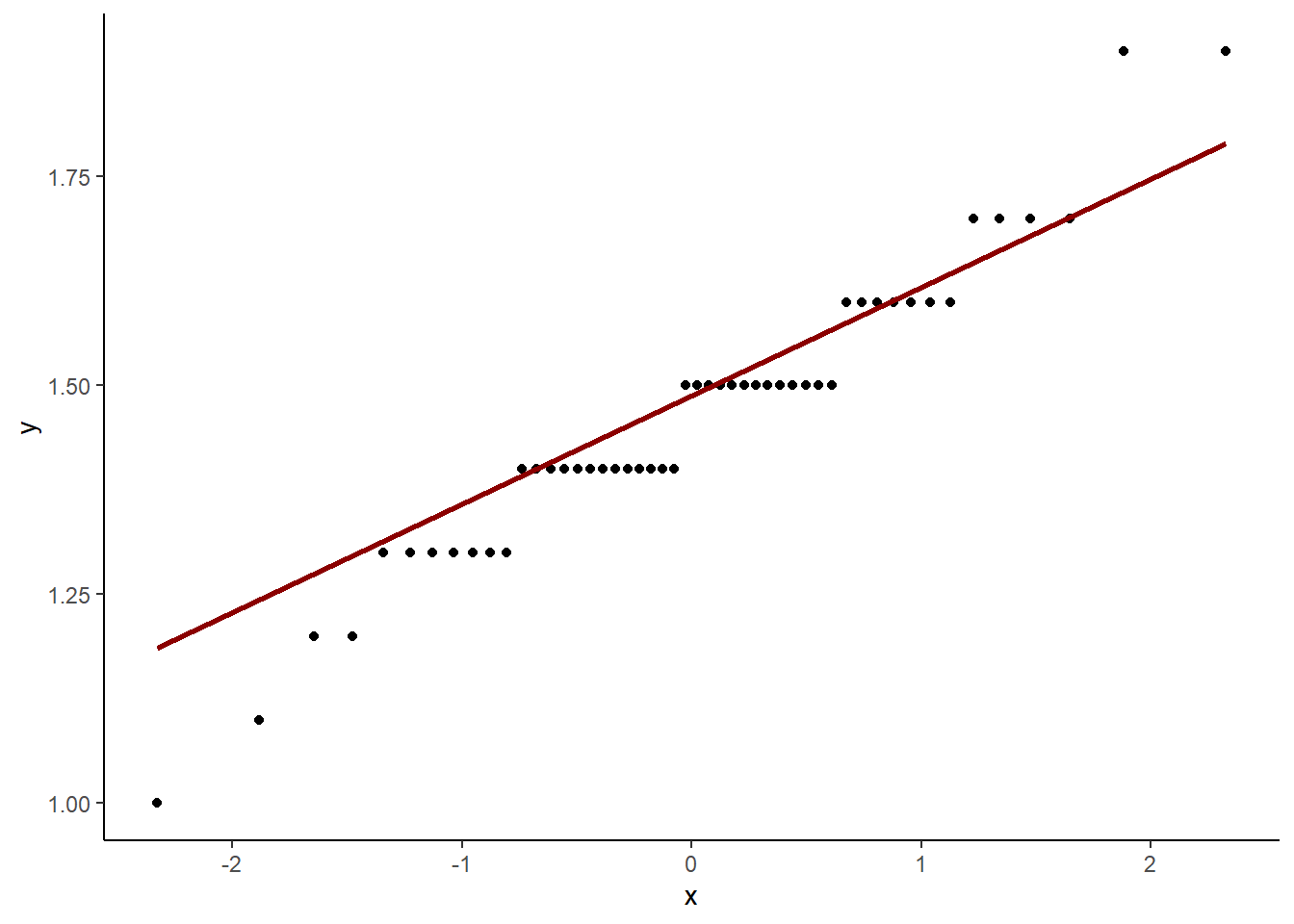
iris %>%
filter(Species == "versicolor") %>%
ggplot() +
aes(sample = Petal.Length) +
geom_qq() +
geom_qq_line() +
theme_classic()