library(tidyverse)Dernière exploration du {tidyverse} avec le package {purrr}
R-Ladies Paris
R-Ladies Paris est une section locale de l’organisation mondiale R-Ladies Global qui promeut la diversité dans la communauté R et fournit un réseau de soutien convivial pour les passionnés du langage de programmation R dans le monde entier.
Introduction
Historique
Le {tidyverse} s’appelait encore le hadleyverse il y a quelques année, c’est-à-dire l'univers de Hadley pour Hadley Wickham son génial créateur.
Le but de Hadley est de rendre l’analyse données plus facile, plus rapide et surtout plus fun et je trouve que cela transparaît dans ses packages !
Le {tidyverse} c’est l’ensemble des packages open-source développé par Hadley et son équipe (Hadley travaille maintenant pour RStudio en plus de plusieurs universités) qui partagent la même philosophie, la même structure de données (le fameux format tidy) et la même syntaxe.
Le format tidy
Le format tidyrepose sur la répétition des lignes des individus afin de limiter le nombre de colonnes.
Dans le plus stricte cas, le format tidy ne présente que 3 colonnes :
_ Identification de l’individu, par exemple : nom_du_pays, num_bague_identification,…
_ Variables mesurées, par exemple : variable peut prendre comme modalités superficie, taille_population, PIB ou masse, taille, longueur_du_bec…
_ Valeur de la mesure. Attention, le format tidy ne supporte par plusieurs type de données dans la même colonne !
La syntaxe tidyverse
Non détaillée ici, je vous invite à consulter le tidyverse style guide.
Les packages concernés
_ ggplot2 : Visulisation des données
_ dplyr : Manipulation des données (filtrer, trier,…) à ne pas confondre avec tidyr qui manipule le format du jeu de données. Présenté le 7 février sur twitch.
_ tidyr : Modification du format du jeu de données pour en faire un jeu de donnée tidy. Présenté le 7 février sur twitch.
_ readr : Lecture rapide de fichiers de données format csv et autres. Attention : format xslx non pris en charge, il faut utiliser le package readxl qui fait partie du tidyverse au sens large mais qui n’est pas attaché par défaut quand on fait library(tidyverse)
_ purrr : Permet le remplacement d’un grand nombre de boucles Vu aujourd’hui
_ tibble : Format des données tidy
_ stringr : Manipulation des chaînes de caractères. Présenté le 21 mars sur twitch.
_ forcats : Manipulation des variables facteurs factors. Présenté le 21 mars sur twitch.
_ lubrdate : Manipulation des dates. Nouveau dans le {tidyverse}
{purrr}
La différence avec les boucles
Elle peut-être réalisée avec une boucle for(), if() ou while() ou en utilisant la vectorisation.
Lors d’une boucle, les vérifications sont faites à chaque itération ce qui peut beaucoup augmenter le temps de calcul.
Il est aussi nécessaire d’initié l’objet contrairement à l’utilisation des vecteurs.
Exemple : création d’un objet contenant les doubles de 1 à 10
objet <- 2
for (i in (2:10)){
objet[i] <- i*2
}Le même objet peut être créé avec la vectorisation qui est la base dans R.
vecteur <- (1:10)*2
identical(objet, vecteur)[1] TRUEPremier exemple d’utilisation de {purrr} avec du texte
Utilisation du début du livre, Les mémoires d’Hadrien de Marguerite Yourcenar.
hadrien <- "Je suis descendu ce matin chez mon médecin Hermogène, qui vient de rentrer à la Villa après un assez long voyage en Asie. L'examen devait se faire à jeun : nous avions pris rendez-vous pour les premières heures de la matinée. Je me suis couché sur un lit après m'être dépouillé de mon manteau et de ma tunique. Je t'épargne des détails qui te seraient aussi désagréables qu'à moimême, et la description du corps d'un homme qui avance en âge et s'apprête à mourir d'une hydropisie du cœur."
# séparer les phrases
paragraphe <- str_split(hadrien, "\\.")
# afficher la longueur
str_length(paragraphe)[1] 506# totale
map(.x = paragraphe, .f = ~ str_length(.x))[[1]]
[1] 120 103 84 177 0# beaucoup de fonctions sont prévue pour la vectorisation et peuvent foncitonner sans mapping
hadrien %>%
str_split("\\.", simplify = TRUE) %>%
str_length()[1] 120 103 84 177 0# détacher par phrases et par mots
str_split(hadrien, " ", simplify = TRUE) %>% length()[1] 88str_split(paragraphe, " ", simplify = TRUE) %>% length()[1] 92str_split(paragraphe, " ", simplify = TRUE) [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8]
[1,] "c(\"Je" "suis" "descendu" "ce" "matin" "chez" "mon" "médecin"
[,9] [,10] [,11] [,12] [,13] [,14] [,15] [,16] [,17]
[1,] "Hermogène," "qui" "vient" "de" "rentrer" "à" "la" "Villa" "après"
[,18] [,19] [,20] [,21] [,22] [,23] [,24] [,25] [,26]
[1,] "un" "assez" "long" "voyage" "en" "Asie\"," "\"" "L'examen" "devait"
[,27] [,28] [,29] [,30] [,31] [,32] [,33] [,34] [,35]
[1,] "se" "faire" "à" "jeun" ":" "nous" "avions" "pris" "rendez-vous"
[,36] [,37] [,38] [,39] [,40] [,41] [,42] [,43] [,44]
[1,] "pour" "les" "premières" "heures" "de" "la" "matinée\"," "\"" "Je"
[,45] [,46] [,47] [,48] [,49] [,50] [,51] [,52] [,53] [,54]
[1,] "me" "suis" "couché" "sur" "un" "lit" "après" "m'être" "dépouillé" "de"
[,55] [,56] [,57] [,58] [,59] [,60] [,61] [,62] [,63]
[1,] "mon" "manteau" "et" "de" "ma" "tunique\"," "\"" "Je" "t'épargne"
[,64] [,65] [,66] [,67] [,68] [,69] [,70] [,71]
[1,] "des" "détails" "qui" "te" "seraient" "aussi" "désagréables" "qu'à"
[,72] [,73] [,74] [,75] [,76] [,77] [,78] [,79] [,80]
[1,] "moimême," "et" "la" "description" "du" "corps" "d'un" "homme" "qui"
[,81] [,82] [,83] [,84] [,85] [,86] [,87] [,88] [,89]
[1,] "avance" "en" "âge" "et" "s'apprête" "à" "mourir" "d'une" "hydropisie"
[,90] [,91] [,92]
[1,] "du" "cœur\"," "\n\"\")"# changer de profondeur dans l'objet grâce à `_depth`
map(
.x = paragraphe,
.f = ~ str_split(.x, " ")
) %>%
map_depth(2, length)[[1]]
[[1]][[1]]
[1] 23
[[1]][[2]]
[1] 19
[[1]][[3]]
[1] 18
[[1]][[4]]
[1] 31
[[1]][[5]]
[1] 1# utilisation de `map2()` lorsque deux listes sont intégrée et `pmap()` lorsqu'il y a plus de deux listes
map2_chr(
.x = c("Alice", "Marc", "Julie", "Charlie"),
.y = c("plage", "montagne", "campagne", "cuisine"),
.f = ~ str_c(.x, " va à la ", .y)
)[1] "Alice va à la plage" "Marc va à la montagne"
[3] "Julie va à la campagne" "Charlie va à la cuisine"# type d'objet produit par map -> une liste par défaut
map2(
.x = c("Alice", "Marc", "Julie", "Charlie"),
.y = c("plage", "montagne", "campagne", "cuisine"),
~ str_c(.x, " va à la ", .y)
) %>%
class()[1] "list"# possibilité de changer le type de sortie avec _chr ou _dbl...
map2_chr(
.x = c("Alice", "Marc", "Julie", "Charlie"),
.y = c("plage", "montagne", "campagne", "cuisine"),
~ str_c(.x, " va à la ", .y)
) %>%
class()[1] "character"map2_chr(
.x = c("Alice", "Marc", "Julie", "Charlie"),
.y = c("plage", "montagne", "campagne", "cuisine"),
~ str_c(.x, " va à la ", .y)
)[1] "Alice va à la plage" "Marc va à la montagne"
[3] "Julie va à la campagne" "Charlie va à la cuisine"Deuxième exemple avec une fonction sur des jdd
Commençons par voir la fonction across() du package {dplyr} qui permet d’applique une ou plusieurs fonctions sur des colonnes multiples.
# calcul des moyennes de sepal_length
iris %>%
summarise(sepal_length_moyenne = mean(Sepal.Length)) sepal_length_moyenne
1 5.843333# application à toutes les colonnes numériques
iris %>%
summarise(
across(
.cols = where(is.numeric),
.fns = list(moyenne = ~ mean(.x, na.rm = TRUE), minimum = min, maximum = max),
.names = "{col}_{fn}"
)
) Sepal.Length_moyenne Sepal.Length_minimum Sepal.Length_maximum
1 5.843333 4.3 7.9
Sepal.Width_moyenne Sepal.Width_minimum Sepal.Width_maximum
1 3.057333 2 4.4
Petal.Length_moyenne Petal.Length_minimum Petal.Length_maximum
1 3.758 1 6.9
Petal.Width_moyenne Petal.Width_minimum Petal.Width_maximum
1 1.199333 0.1 2.5Réalisation sur différentes tables de données grâce à la fonction map() de {purrr}.
# travail sur les jdd de données iris, mtcars et women
map(
.x = list(iris, mtcars, women),
.f = ~ summarise(
.x,
across(
.cols = where(is.numeric),
.fns = list(moyenne = ~ mean(.x, na.rm = TRUE), minimum = min, maximum = max),
.names = "{col}_{fn}"
)
)
)[[1]]
Sepal.Length_moyenne Sepal.Length_minimum Sepal.Length_maximum
1 5.843333 4.3 7.9
Sepal.Width_moyenne Sepal.Width_minimum Sepal.Width_maximum
1 3.057333 2 4.4
Petal.Length_moyenne Petal.Length_minimum Petal.Length_maximum
1 3.758 1 6.9
Petal.Width_moyenne Petal.Width_minimum Petal.Width_maximum
1 1.199333 0.1 2.5
[[2]]
mpg_moyenne mpg_minimum mpg_maximum cyl_moyenne cyl_minimum cyl_maximum
1 20.09062 10.4 33.9 6.1875 4 8
disp_moyenne disp_minimum disp_maximum hp_moyenne hp_minimum hp_maximum
1 230.7219 71.1 472 146.6875 52 335
drat_moyenne drat_minimum drat_maximum wt_moyenne wt_minimum wt_maximum
1 3.596563 2.76 4.93 3.21725 1.513 5.424
qsec_moyenne qsec_minimum qsec_maximum vs_moyenne vs_minimum vs_maximum
1 17.84875 14.5 22.9 0.4375 0 1
am_moyenne am_minimum am_maximum gear_moyenne gear_minimum gear_maximum
1 0.40625 0 1 3.6875 3 5
carb_moyenne carb_minimum carb_maximum
1 2.8125 1 8
[[3]]
height_moyenne height_minimum height_maximum weight_moyenne weight_minimum
1 65 58 72 136.7333 115
weight_maximum
1 164# ajout des noms des jeux de données
map(
.x = list(iris, mtcars, women),
.f = ~ summarise(
.x,
across(
.cols = where(is.numeric),
.fns = list(moyenne = ~ mean(.x, na.rm = TRUE), minimum = min, maximum = max),
.names = "{col}_{fn}"
)
)
) %>%
set_names(c("iris", "mtcars", "women"))$iris
Sepal.Length_moyenne Sepal.Length_minimum Sepal.Length_maximum
1 5.843333 4.3 7.9
Sepal.Width_moyenne Sepal.Width_minimum Sepal.Width_maximum
1 3.057333 2 4.4
Petal.Length_moyenne Petal.Length_minimum Petal.Length_maximum
1 3.758 1 6.9
Petal.Width_moyenne Petal.Width_minimum Petal.Width_maximum
1 1.199333 0.1 2.5
$mtcars
mpg_moyenne mpg_minimum mpg_maximum cyl_moyenne cyl_minimum cyl_maximum
1 20.09062 10.4 33.9 6.1875 4 8
disp_moyenne disp_minimum disp_maximum hp_moyenne hp_minimum hp_maximum
1 230.7219 71.1 472 146.6875 52 335
drat_moyenne drat_minimum drat_maximum wt_moyenne wt_minimum wt_maximum
1 3.596563 2.76 4.93 3.21725 1.513 5.424
qsec_moyenne qsec_minimum qsec_maximum vs_moyenne vs_minimum vs_maximum
1 17.84875 14.5 22.9 0.4375 0 1
am_moyenne am_minimum am_maximum gear_moyenne gear_minimum gear_maximum
1 0.40625 0 1 3.6875 3 5
carb_moyenne carb_minimum carb_maximum
1 2.8125 1 8
$women
height_moyenne height_minimum height_maximum weight_moyenne weight_minimum
1 65 58 72 136.7333 115
weight_maximum
1 164# format tableau croisé
map(
.x = list(iris, mtcars, women),
.f = ~ summarise(
.x,
across(
.cols = where(is.numeric),
.fns = list(moyenne = ~ mean(.x, na.rm = TRUE), minimum = min, maximum = max),
.names = "{col}_{fn}"
)
)
) %>%
set_names(c("iris", "mtcars", "women")) %>%
map(
.f = ~ pivot_longer(
.x,
cols = everything(),
names_to = c("mesure", ".value"),
names_pattern = "(.+)_(.+)")
)$iris
# A tibble: 4 × 4
mesure moyenne minimum maximum
<chr> <dbl> <dbl> <dbl>
1 Sepal.Length 5.84 4.3 7.9
2 Sepal.Width 3.06 2 4.4
3 Petal.Length 3.76 1 6.9
4 Petal.Width 1.20 0.1 2.5
$mtcars
# A tibble: 11 × 4
mesure moyenne minimum maximum
<chr> <dbl> <dbl> <dbl>
1 mpg 20.1 10.4 33.9
2 cyl 6.19 4 8
3 disp 231. 71.1 472
4 hp 147. 52 335
5 drat 3.60 2.76 4.93
6 wt 3.22 1.51 5.42
7 qsec 17.8 14.5 22.9
8 vs 0.438 0 1
9 am 0.406 0 1
10 gear 3.69 3 5
11 carb 2.81 1 8
$women
# A tibble: 2 × 4
mesure moyenne minimum maximum
<chr> <dbl> <dbl> <dbl>
1 height 65 58 72
2 weight 137. 115 164Troisième exemple avec le traitement de colonnes d’un même jdd
Exemple : avec les données {ascombes}
C’est un jeu de données de “4” groupes pour lesquelles les propriétés statistiques simple (moyennes, variance, corrélation, régression linéaire) sont similaire alors que les données sont très différentes.
anscombe x1 x2 x3 x4 y1 y2 y3 y4
1 10 10 10 8 8.04 9.14 7.46 6.58
2 8 8 8 8 6.95 8.14 6.77 5.76
3 13 13 13 8 7.58 8.74 12.74 7.71
4 9 9 9 8 8.81 8.77 7.11 8.84
5 11 11 11 8 8.33 9.26 7.81 8.47
6 14 14 14 8 9.96 8.10 8.84 7.04
7 6 6 6 8 7.24 6.13 6.08 5.25
8 4 4 4 19 4.26 3.10 5.39 12.50
9 12 12 12 8 10.84 9.13 8.15 5.56
10 7 7 7 8 4.82 7.26 6.42 7.91
11 5 5 5 8 5.68 4.74 5.73 6.89# réalisation d'une régression linéaire
## simple
lm(y1 ~ x1, data = anscombe) %>%
summary()
Call:
lm(formula = y1 ~ x1, data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.92127 -0.45577 -0.04136 0.70941 1.83882
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0001 1.1247 2.667 0.02573 *
x1 0.5001 0.1179 4.241 0.00217 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.237 on 9 degrees of freedom
Multiple R-squared: 0.6665, Adjusted R-squared: 0.6295
F-statistic: 17.99 on 1 and 9 DF, p-value: 0.00217## sur toutes les colonnes
map2(
.x = c("x1", "x2", "x3", "x4"),
.y = c("y1", "y2", "y3", "y4"),
.f = ~ lm(get(.y) ~ get(.x), data = anscombe) %>% summary()
)[[1]]
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.92127 -0.45577 -0.04136 0.70941 1.83882
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0001 1.1247 2.667 0.02573 *
get(.x) 0.5001 0.1179 4.241 0.00217 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.237 on 9 degrees of freedom
Multiple R-squared: 0.6665, Adjusted R-squared: 0.6295
F-statistic: 17.99 on 1 and 9 DF, p-value: 0.00217
[[2]]
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.9009 -0.7609 0.1291 0.9491 1.2691
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.001 1.125 2.667 0.02576 *
get(.x) 0.500 0.118 4.239 0.00218 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.237 on 9 degrees of freedom
Multiple R-squared: 0.6662, Adjusted R-squared: 0.6292
F-statistic: 17.97 on 1 and 9 DF, p-value: 0.002179
[[3]]
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.1586 -0.6146 -0.2303 0.1540 3.2411
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0025 1.1245 2.670 0.02562 *
get(.x) 0.4997 0.1179 4.239 0.00218 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.236 on 9 degrees of freedom
Multiple R-squared: 0.6663, Adjusted R-squared: 0.6292
F-statistic: 17.97 on 1 and 9 DF, p-value: 0.002176
[[4]]
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.751 -0.831 0.000 0.809 1.839
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0017 1.1239 2.671 0.02559 *
get(.x) 0.4999 0.1178 4.243 0.00216 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.236 on 9 degrees of freedom
Multiple R-squared: 0.6667, Adjusted R-squared: 0.6297
F-statistic: 18 on 1 and 9 DF, p-value: 0.002165## avec l'utilisation de la fonction glue
library(glue)
library(glue)
glue("x{1:4}")x1
x2
x3
x4## utilisation de set_names() pour le nom des jdd
map2(
.x = c(glue("x{1:4}")),
.y = c(glue("y{1:4}")),
.f = ~ lm(get(.y) ~ get(.x), data = anscombe) %>% summary()
) %>%
set_names(glue("colonnes finissants par {1:4}"))$`colonnes finissants par 1`
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.92127 -0.45577 -0.04136 0.70941 1.83882
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0001 1.1247 2.667 0.02573 *
get(.x) 0.5001 0.1179 4.241 0.00217 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.237 on 9 degrees of freedom
Multiple R-squared: 0.6665, Adjusted R-squared: 0.6295
F-statistic: 17.99 on 1 and 9 DF, p-value: 0.00217
$`colonnes finissants par 2`
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.9009 -0.7609 0.1291 0.9491 1.2691
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.001 1.125 2.667 0.02576 *
get(.x) 0.500 0.118 4.239 0.00218 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.237 on 9 degrees of freedom
Multiple R-squared: 0.6662, Adjusted R-squared: 0.6292
F-statistic: 17.97 on 1 and 9 DF, p-value: 0.002179
$`colonnes finissants par 3`
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.1586 -0.6146 -0.2303 0.1540 3.2411
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0025 1.1245 2.670 0.02562 *
get(.x) 0.4997 0.1179 4.239 0.00218 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.236 on 9 degrees of freedom
Multiple R-squared: 0.6663, Adjusted R-squared: 0.6292
F-statistic: 17.97 on 1 and 9 DF, p-value: 0.002176
$`colonnes finissants par 4`
Call:
lm(formula = get(.y) ~ get(.x), data = anscombe)
Residuals:
Min 1Q Median 3Q Max
-1.751 -0.831 0.000 0.809 1.839
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0017 1.1239 2.671 0.02559 *
get(.x) 0.4999 0.1178 4.243 0.00216 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.236 on 9 degrees of freedom
Multiple R-squared: 0.6667, Adjusted R-squared: 0.6297
F-statistic: 18 on 1 and 9 DF, p-value: 0.002165# vérification grâce à un graphique
ggplot(anscombe) +
aes(x = x1, y = y1) +
geom_point() +
geom_abline(slope = 0.5, intercept = 3, color = "blue") +
theme_classic()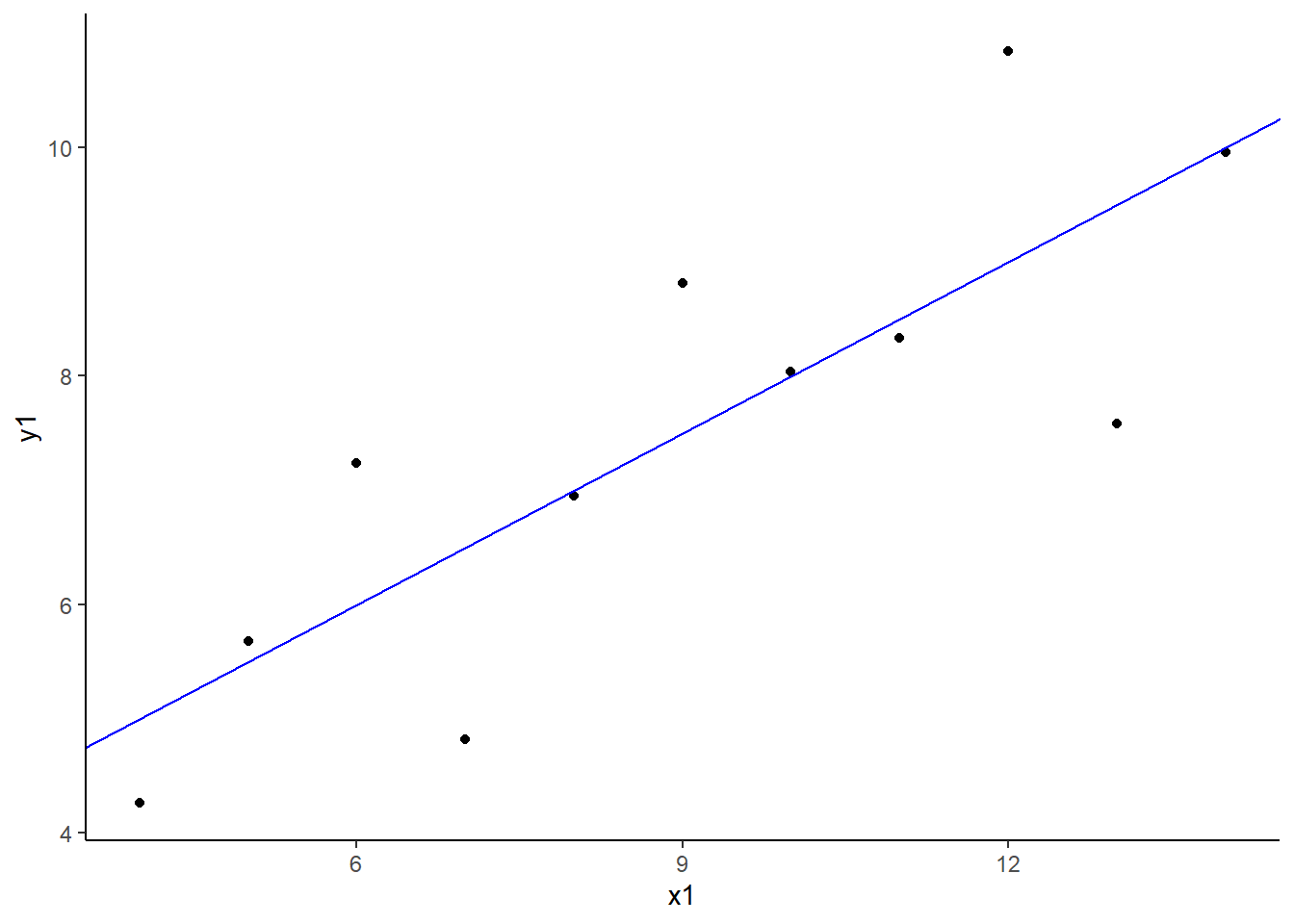
## sur toutes les colonnes
purrr::map2(
.x = c(glue("x{1:4}")),
.y = c(glue("y{1:4}")),
.f = ~ ggplot(anscombe) +
aes(x = get(.x), y = get(.y)) +
geom_point() +
geom_abline(slope = 0.5, intercept = 3, color = "blue") +
theme_classic()
)[[1]]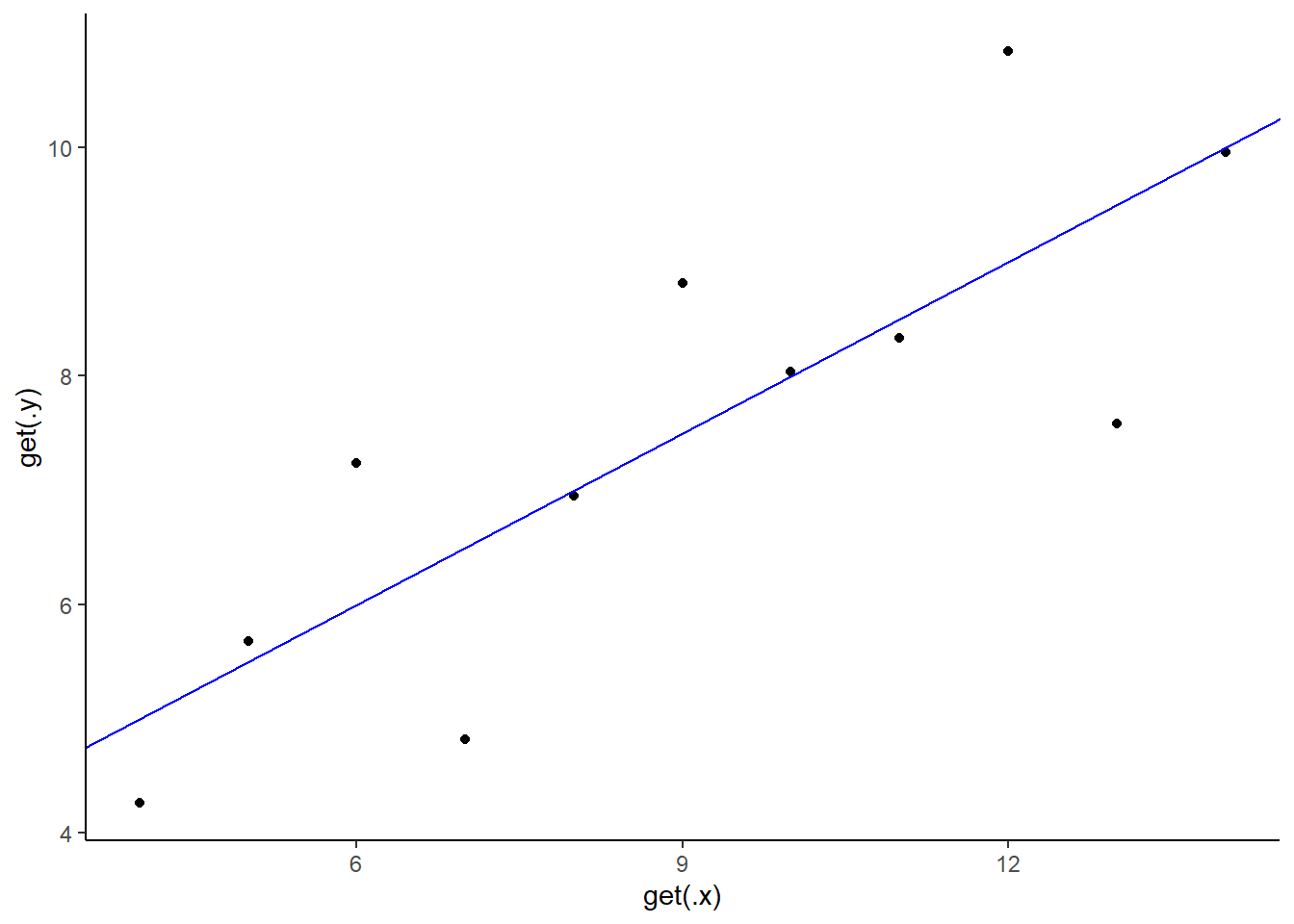
[[2]]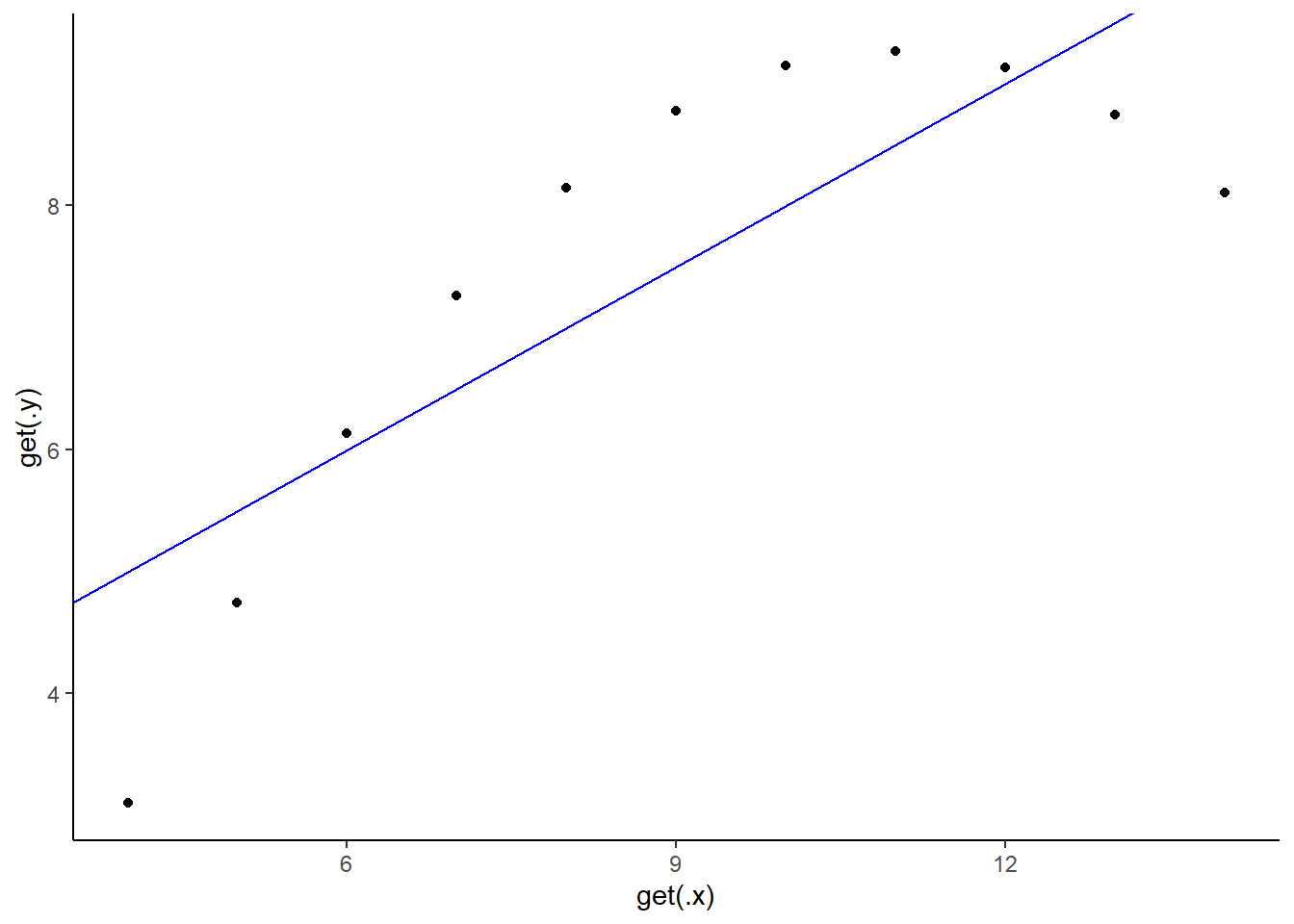
[[3]]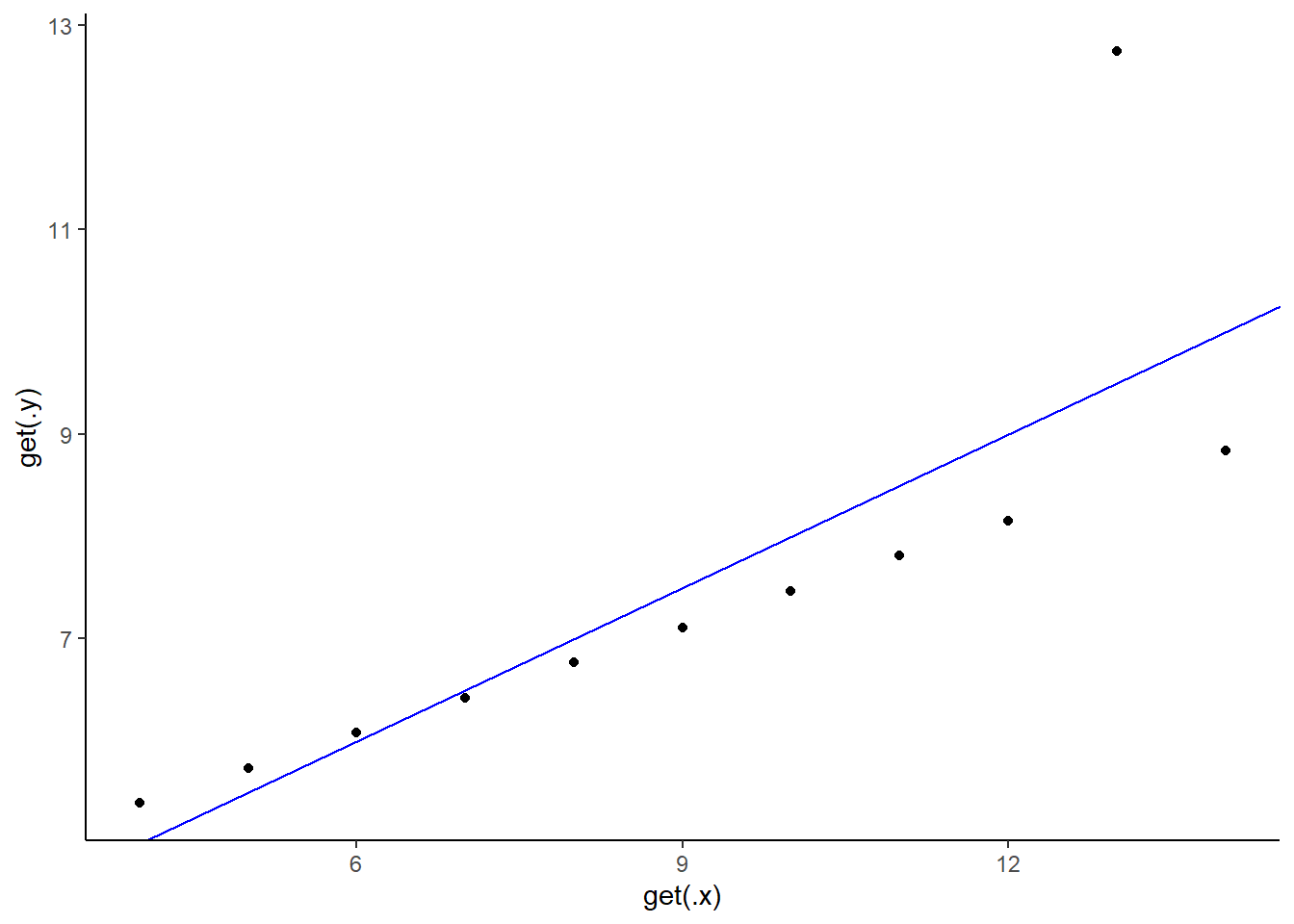
[[4]]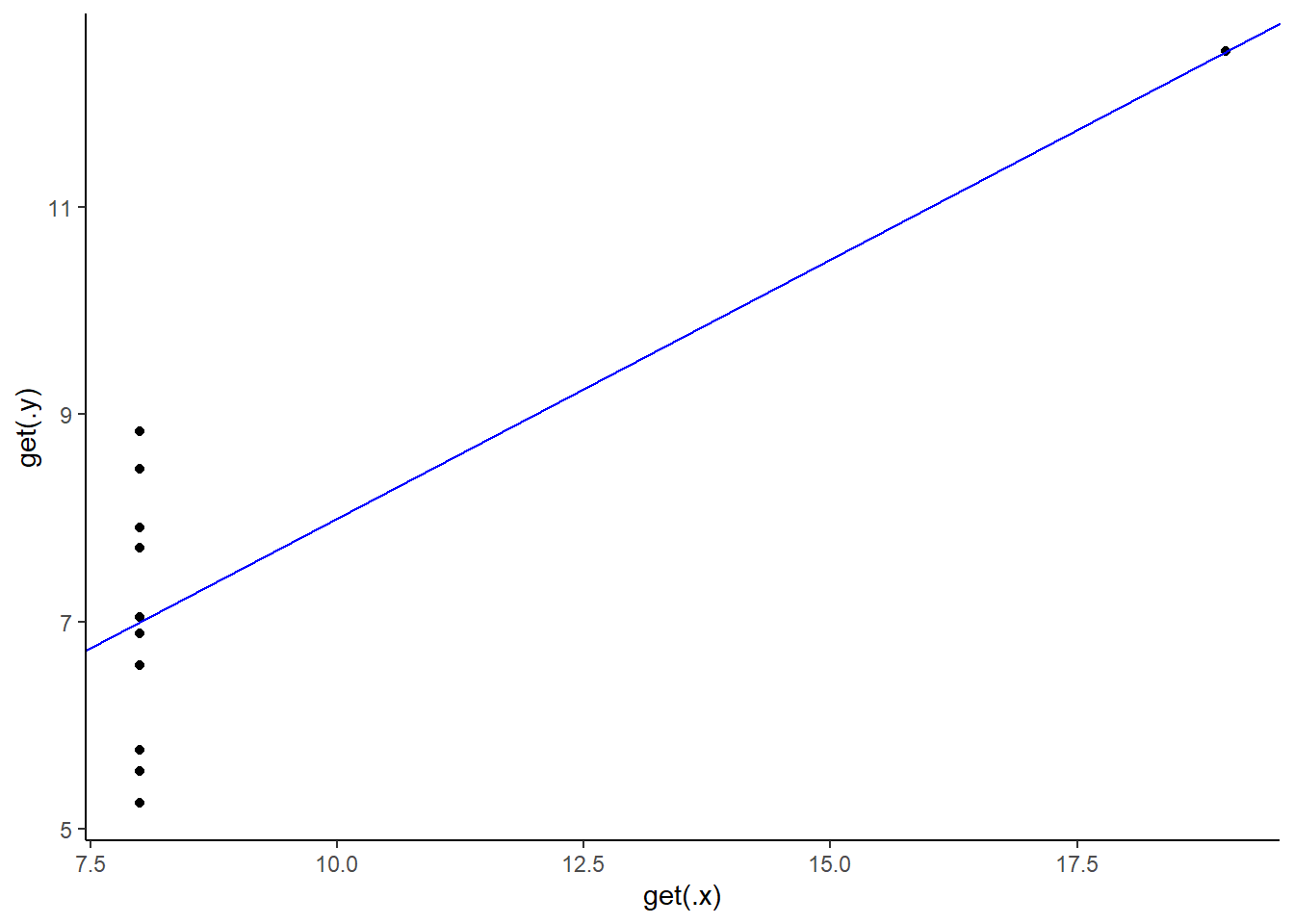
## modification des noms des axes
purrr::map2(
.x = c(glue("x{1:4}")),
.y = c(glue("y{1:4}")),
.f = ~ ggplot(anscombe) +
aes(x = get(.x), y = get(.y)) +
geom_point() +
geom_abline(slope = 0.5, intercept = 3, color = "blue") +
labs(x = .x, y = .y) +
theme_classic()
)[[1]]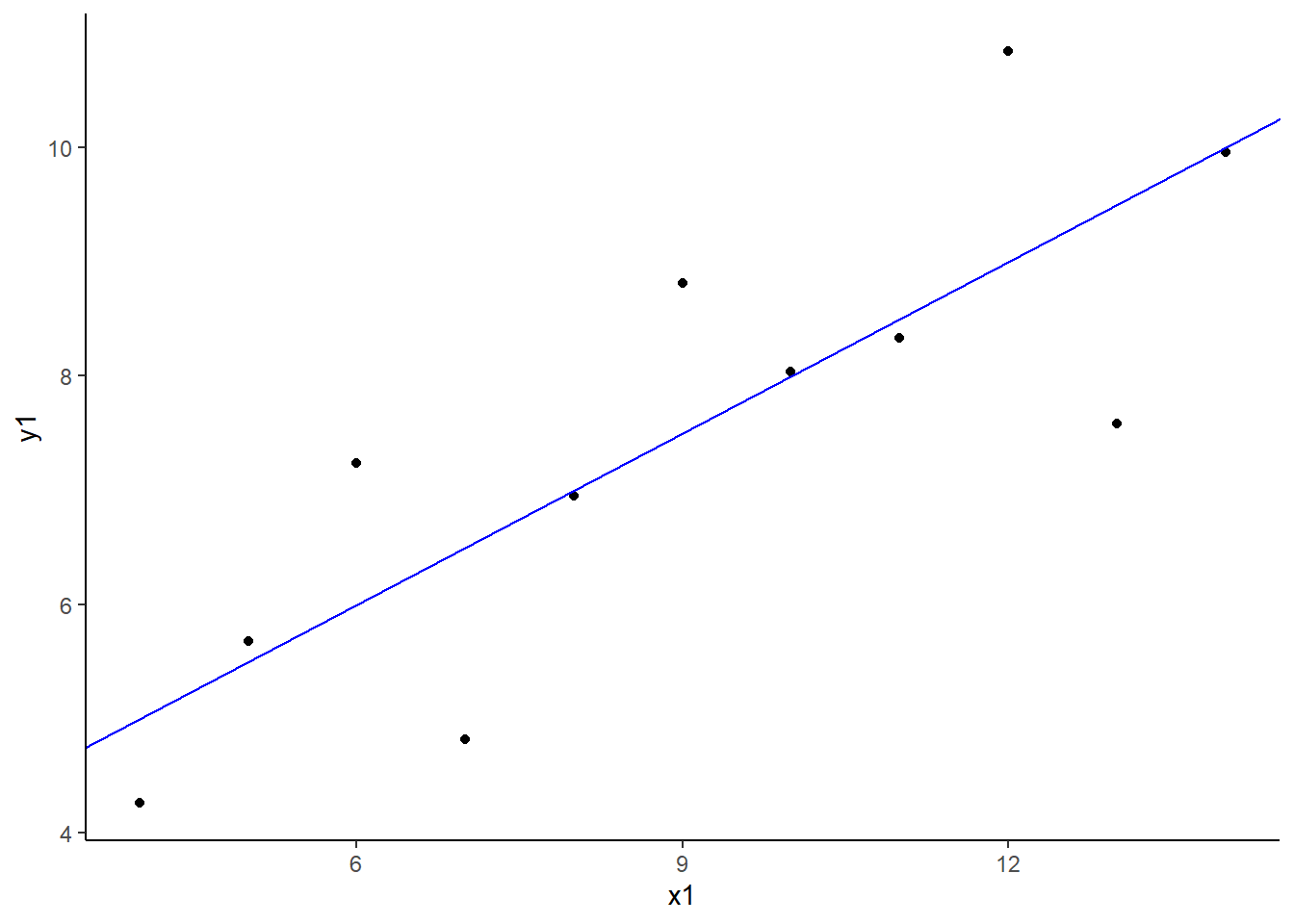
[[2]]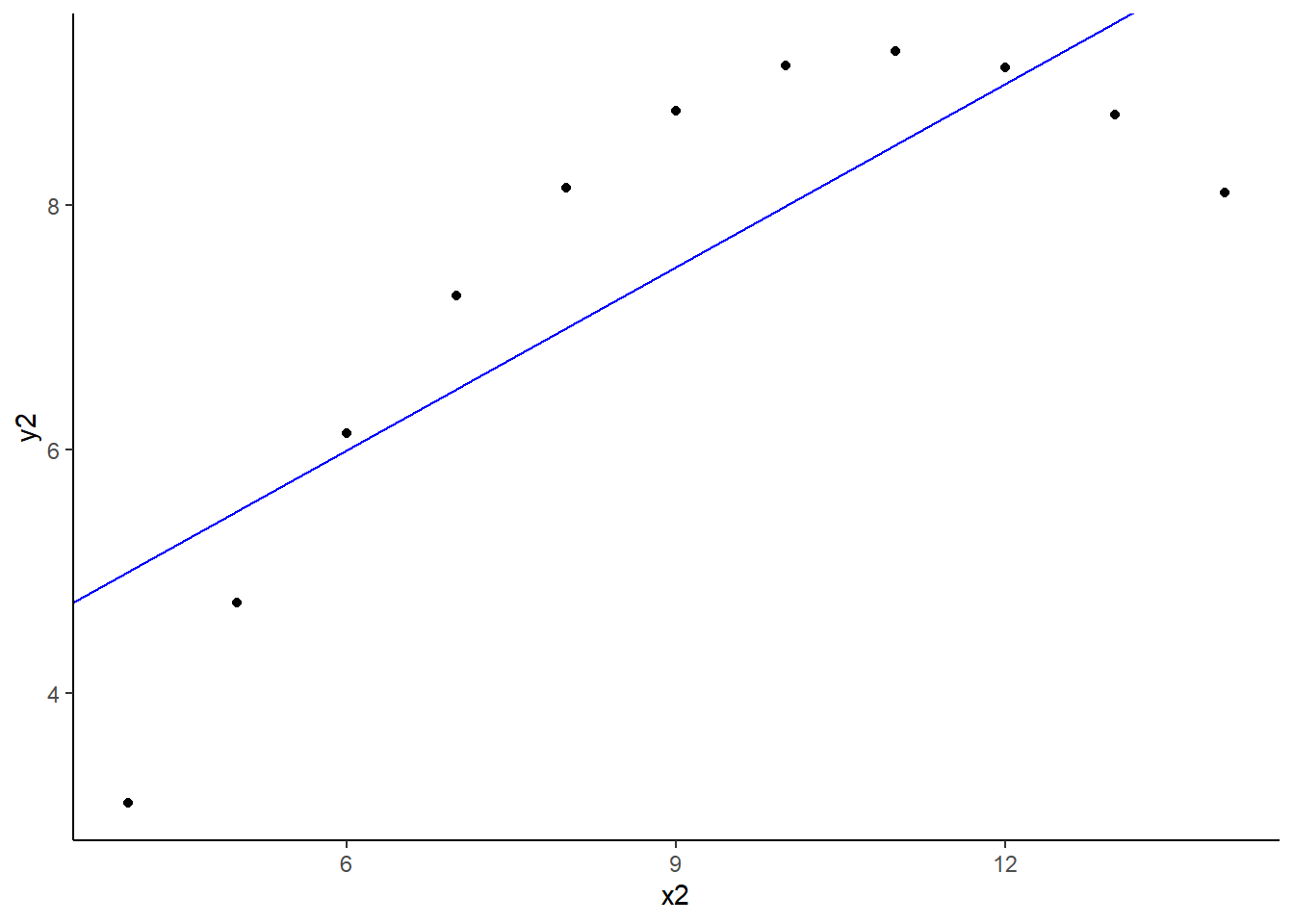
[[3]]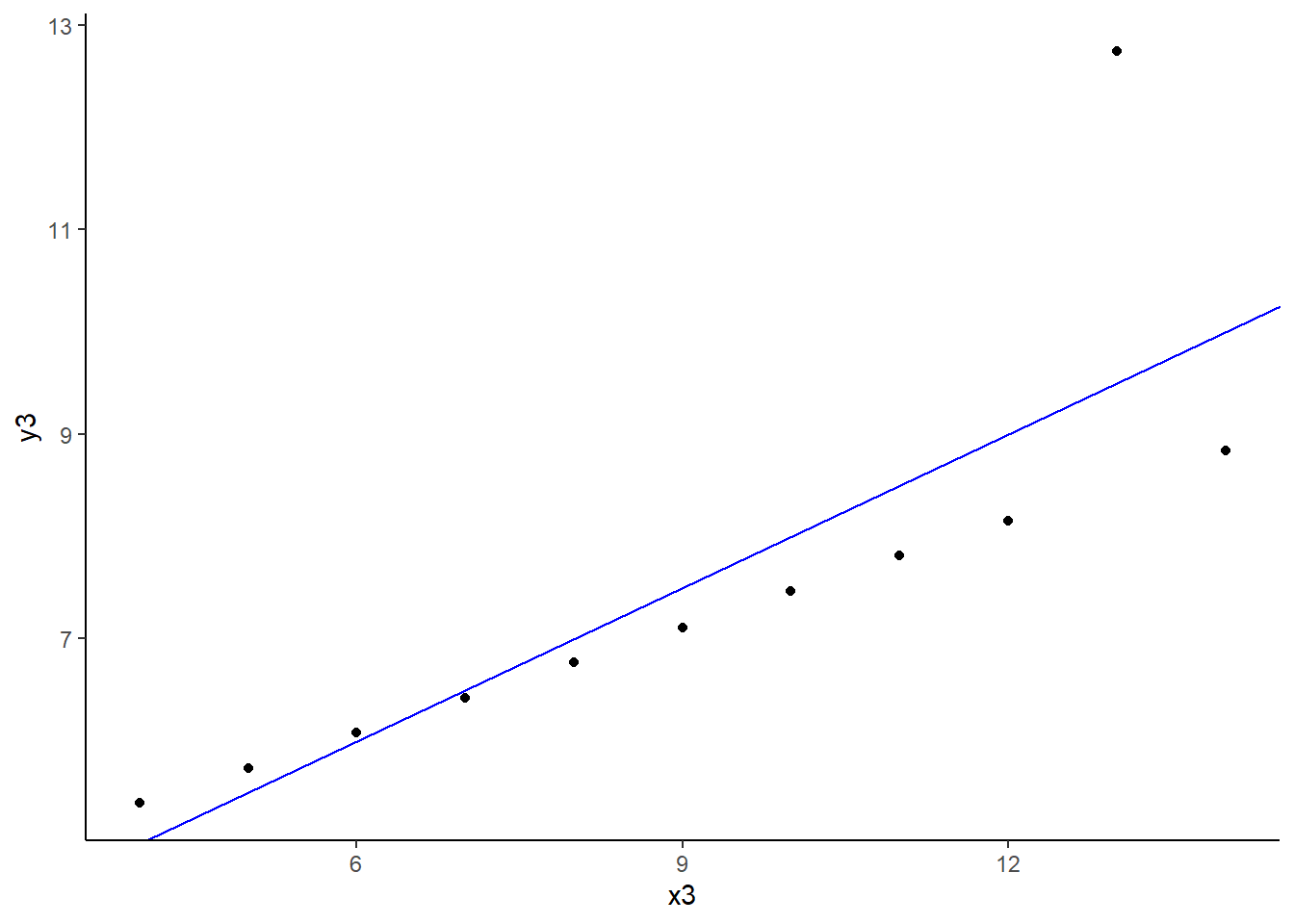
[[4]]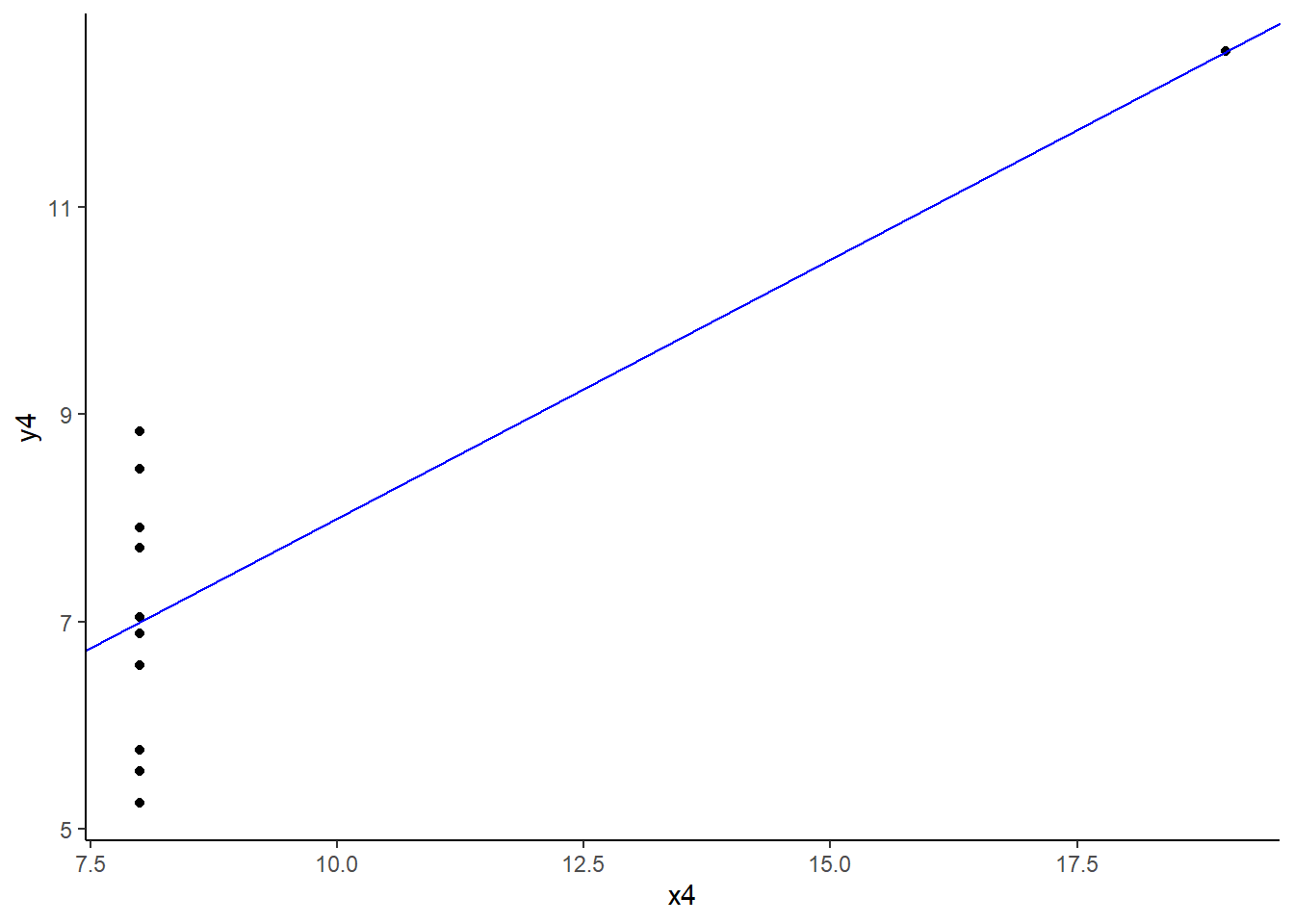
## enregistrement automatique des graphiques générés
purrr::map2(
.x = c(glue("x{1:4}")),
.y = c(glue("y{1:4}")),
.f = ~ ggplot(anscombe) +
aes(x = get(.x), y = get(.y)) +
geom_point() +
geom_abline(slope = 0.5, intercept = 3, color = "blue") +
labs(x = .x, y = .y) +
theme_classic()
) %>%
purrr::map2(
.y = c(glue("img/x{1:4}y{1:4}.png")),
.f = ~ ggsave(plot = .x, filename = .y, width = 2, height = 2)
)[[1]]
[1] "img/x1y1.png"
[[2]]
[1] "img/x2y2.png"
[[3]]
[1] "img/x3y3.png"
[[4]]
[1] "img/x4y4.png"Ressources intéressantes
_ le cheatsheet de {purrr}
_ la vignette de {purrr}
_ La page d’itération dans l’e-book R for Data Science
